csj009的我的研究目标,其果色与csj010及参考组zunla均不同。需要筛选亲本间不同的位点,来设计引物,打开我的目标染色体,发现这个染色体有230多mb,位点大概有1907252个,光突变位点文件就有355mb,那是相当的大,按照导师的要求,每5mb找3个亲本间不同的indel位点,大约就是140个位点,然后设计引物。在近200万个位点中找140个位点,那真的是大海捞针,必须对这个突变信息文件进行过滤筛选。
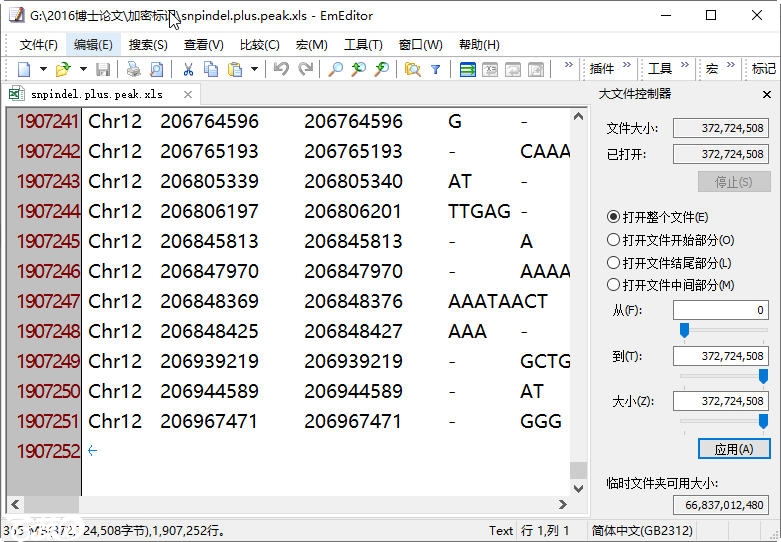
首先用EmEditor打开9号染色体突变体信息文件
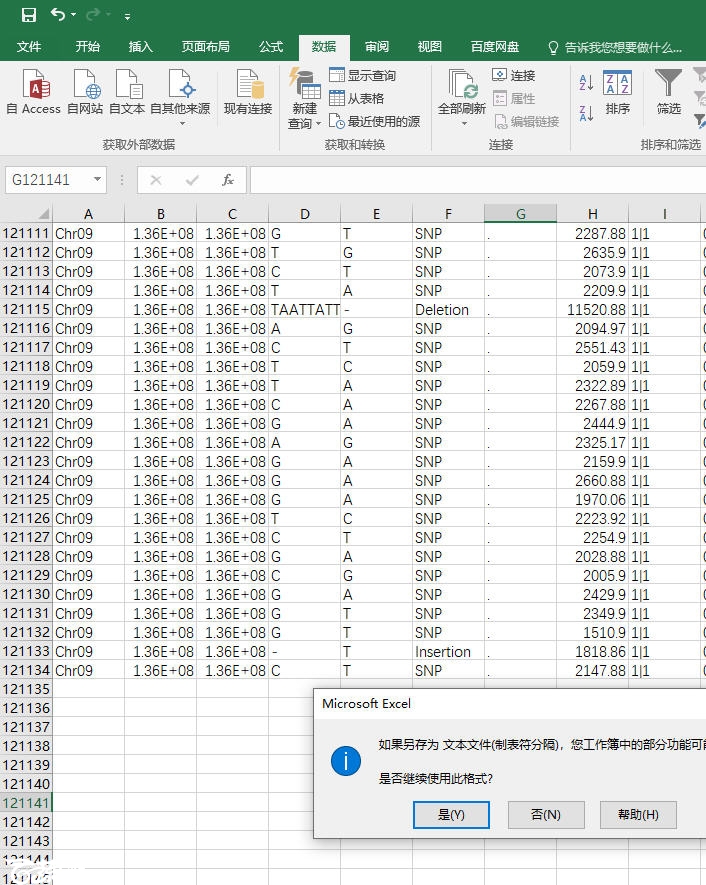
可以看到这个数据有1907252行,超过了excel表最大数据量1048576,那么直接用excel打开肯定不能显示全,那么首先就是分隔,这个简单,就直接从中间截取,80多万行excel是没有压力的,从而把snpindel.plus.peak.xls这个文件分隔为snpindel.plus.peak.1.xls和snpindel.plus.peak.2.xls,然后打开两个文件,分别进行过滤清理。
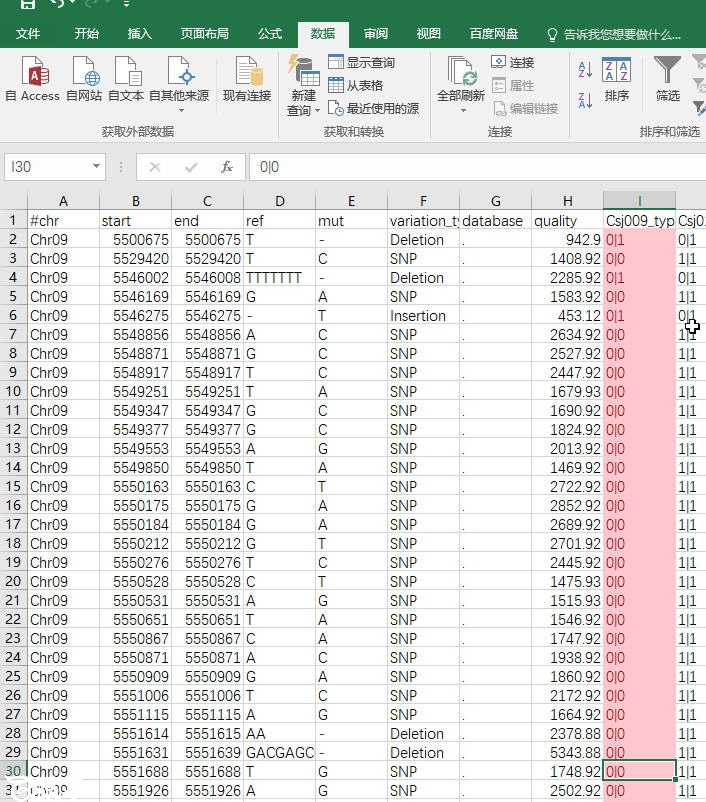
csj009和csj010是我的亲本,那么首先就是剔除亲本中不合格的位点。从图中可以看到有两种,一种是表示“0|1”,代表这个位点是杂合的,另外一种是“.|.”,表明不清楚类型。首先清除亲本中的这两种位点,碱基型中1|1 代表纯合突变,0|1 代表杂合突变,0|0 代表与参考一致,。
这个方法以前介绍过。
1,点选“Csj009_type”,选择菜单中的“条件格式”“突出显示单元格规则”,“等于”,把“0|1”填进去。
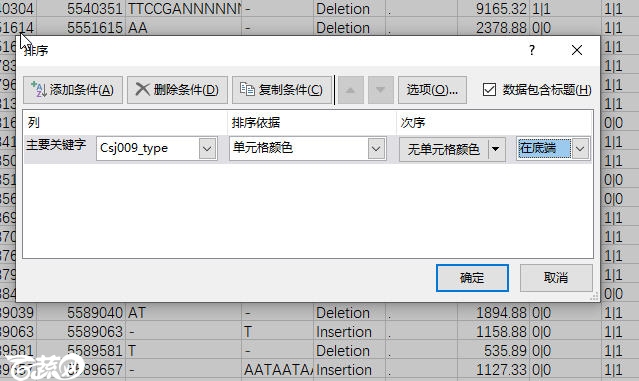
这样就把所有的含有”0|1”的单元标红了。之后选择整个表格,在“数据”中找到“排序”,
按照“主要关键字”的“单元格颜色”无色在低端,有色在顶端,这样排序。
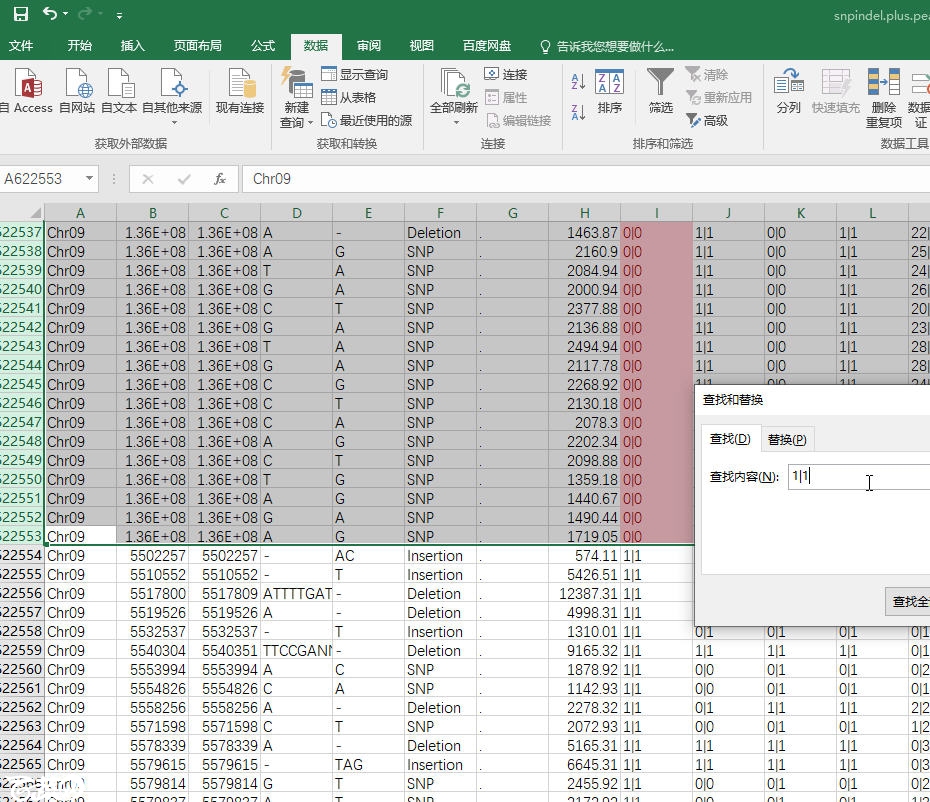
这样就把csj009中杂合位点(0|1),不明位点(.|.)及和参考组一致的位点(0|0)的表格全部标红,并且显示在顶端了。之后选择csj009_type这一行,ctrl+f,查找“1|1”,按住shift键往上拉到第一行,全部删除之。
这样一通下来,snpindel.plus.peak.1.xls还剩下121134个位点,snpindel.plus.peak.2.xls得到93138个位点
然后保存
这里选择以文本文件保存,方便两个大文件的合并。
20多万条数据,excel完全可以处理了,以文本文件的方式合并,快捷,方便。这样得到214271条数据,然后另存为xlsx格式文件,不再保留文本格式文件。
2,导师建议全部设计InDel标记,那么需要把SNP突变清除掉。在“variation_type”标记所有SNP为红色,然后排序,删除之。
这么一顿操作下来之后,近200万个位点,只剩下34280个左右了。
3,按照上面方法删除了Csj009_type中含有“0|1”,“.|.”,“0|0”及SNP位点,但是如果亲本的突变位点一样,那么这个位点只是和参考基因组有差异,亲本间无差异。由于csj009的果色与csj010明显不同,那么需要将亲本间一致的突变清理出去,这个怎么做呢。
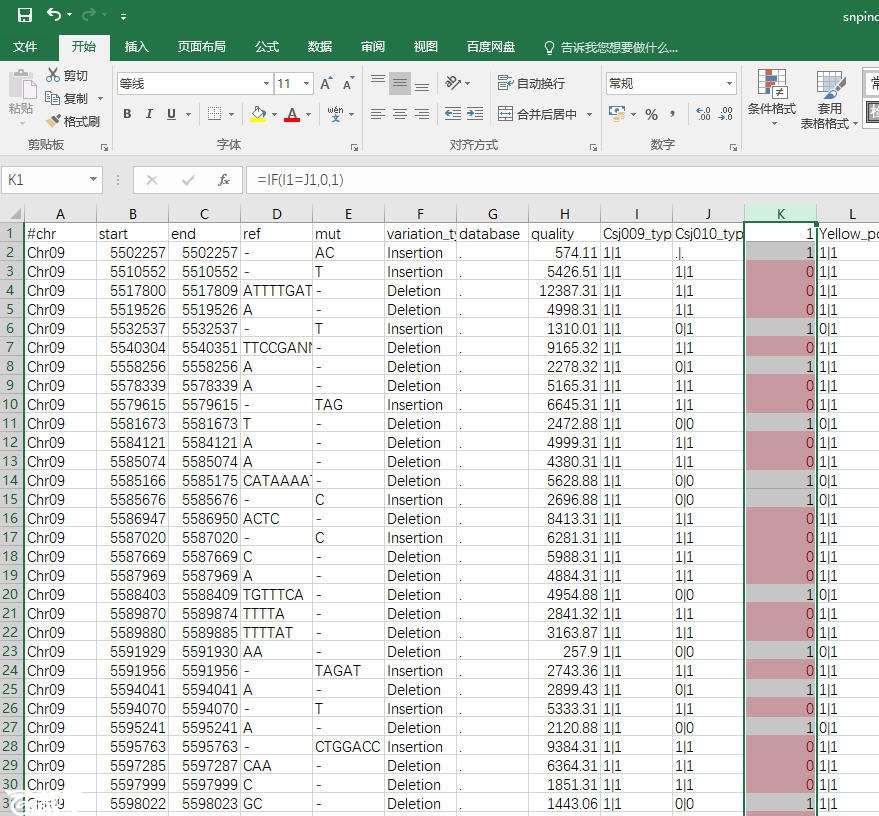
方法也很简单,在“Csj010_type”后面插入新的1列,然后选择之,在命令栏输入命令“=IF(I1=J1,0,1)”,按住ctrl键,然后敲击enter键,那么“Csj009_type”和“Csj010_type”中内容相同的列将为0,不同的将为1.然后按照上面的方法,把0标红,然后筛选,删除就可以了。
然后用排序或筛选(不推荐),就把亲本间一致的突变筛除了,最终得到14665个位点,这就是我们的目标位点区域了,在这里找出我们需要的位点信息,然后设计引物。
转载请注明:百蔬君 » 【原创文章】引物设计中利用excel筛选符合特定要求的位点