这几天折腾snpeff的事情,感觉比annovar好用些。
我得环境是conda,安装snpeff命令
conda install snpeff
然后查找有没有需要的库,我安装的是snpeff4.5
安装指定版本方法
conda install snpeff=4.5
这是目前的最新版,然后列出所有snpeff拥有的数据库,可以保存到txt中、
snpEff databases >snpeff.database.txt
也可以值查找目标数据库,比如我的是辣椒参考组
snpEff databases |grep 'Capsicum' |less -S
最新版是有Capsicum的数据库的,然后下载数据库
snpEff download Capsicum_annuum
但是snpeff抽风,这个数据库无论怎么都下载不下来,不管你是否翻墙。
找了一下具体地址,http://downloads.sourceforge.net/project/snpeff/databases/v4_5/snpEff_v4_5_Capsicum_annuum.zip
确实无法下载,跑到sourceforge.net去看下了,只有v4.3的文件夹,v4.5的文件夹都没有,所有路径都是错误的,这个就不知道原因了。
没办法,只有一条路子了,自建这个Capsicum_annuum的注释库。
我使用的zunla的参考组,这个数据库就叫zunla了。
首先查找snpeff的配置文件位置
find / -name "snpEff.config"
发现有两个,这是因为我使用了conda的环境的原因,一个配置文件是原始的,一个配置文件是环境的,如果使用了环境,那么就修改环境的相关配置文件,并且把相关文件放到环境的相应目录中去。
原始环境
在data下面建立zunla文件夹和genomes文件夹
miniconda2/pkgs/snpeff-4.5covid19-1/share/snpeff-4.5covid19-1/data/zunla
相应配置文件是
miniconda2/pkgs/snpeff-4.5covid19-1/share/snpeff-4.5covid19-1/snpEff.config
自定义环境,我这里环境名是pepper
miniconda2/envs/pepper/share/snpeff-4.5covid19-1/data/zunla
配置文件
miniconda2/envs/pepper/share/snpeff-4.5covid19-1/snpEff.config
在配置文件中添加自定义数据库的命令
#--- # Non-standard Databases #---
下面添加
#zunla zunla.genome : Capsicum_annuum
序列文件名可以命民为zunla.fa或者sequences.fa
放入到genomes或者zunla文件夹均可,genomes文件夹优先读取。
这里需要强调的是必须是.fa扩展名,不认fasta扩展名
注释文件放到建立的zunla的文件中
注释文件命名为
zunla.gff或者genes.gff
gff3文件亦可,准备好之后就可以构建了
snpEff build -gff3 -v zunla
这里必须要注明是gff2还是gff3。注释文件一般三种格式gff3/gff2/-gtf22,扩展名为gtf的肯定是gtf22了,gff3的扩展名一般为gff3。
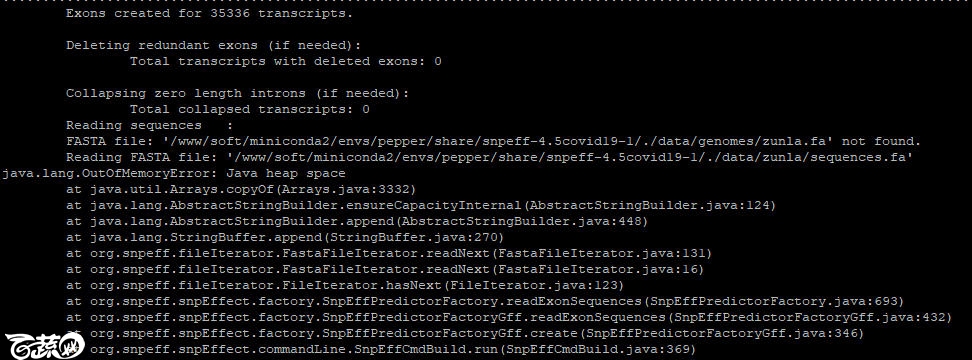
由于辣椒的参考组序列比较大,内存溢出了
修改一下命令
snpEff -Xmx40g build -gff3 -v zunla
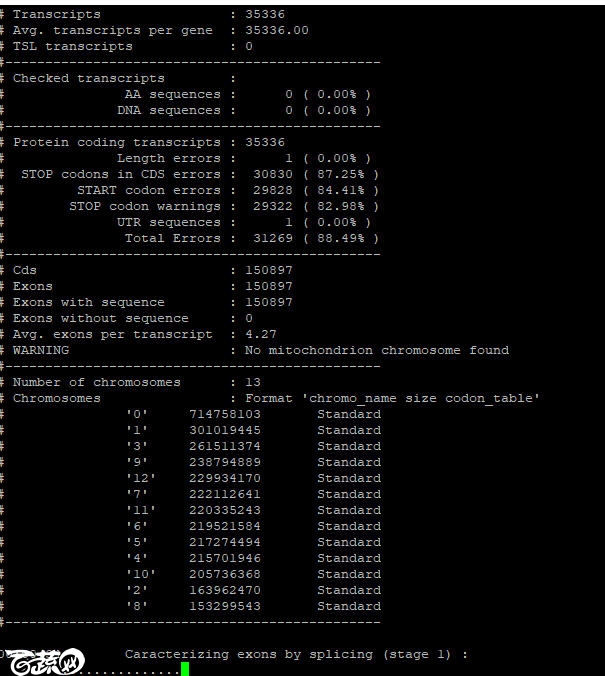
就可以正常构建了
然后再运行注释命令就可以了
snpEff -Xmx40g zunla input.vcf > snpeff.vcf
提醒一点的是所有的snpEff命令中E必须大写!
gff2与gff3格式区别
关于注释文件的格式可以参考这篇文章“常用生物信息学格式介绍(fasta、fastq、gff2、gtf(gff2.5)、gff3、bed、sam、bam、vcf)”
写的比较详细,我仔细研究了一下gff2和gff3之间的区别
可以看出gff2与gff3的区别,关键在于第九列
比如:
Chr1 curated CDS 365647 365963 . + 1 Transcript "R119.7"gff2第九列中属性和键值之间是以空格表示,这段表示该CDS是属于名为R119.7的transcript
在gff3中,他的熟性和键值之间是用等号表示的。
比如
ctg123 . exon 1300 1500 . + . ID=exon00001这里用id=exon00001来表示这个外显子的属性
一般的gff3文件都会在开头注明版本##gff-version 3,但是我的这个注释文件扩展名是gff,开头也没有这个版本的信息。
但gff的文件格式是
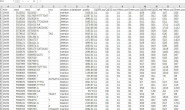
Chr00 GLEAN CDS 191355000 191355087 . + 0 Parent=Capana00g000065;
Chr00 GLEAN CDS 191355173 191355519 . + 2 Parent=Capana00g000065;
Chr00 GLEAN mRNA 191377025 191377768 1 + . ID=Capana00g000066;
Chr00 GLEAN CDS 191377025 191377768 . + 0 Parent=Capana00g000066;
Chr00 GLEAN mRNA 191690806 191691794 1 + . ID=Capana00g000067;
因而我的注释文件是gff3,而不是gff2,这个版本需要查看内容来自己识别。
转载请注明:百蔬君 » 【原创文章】SnpEff自建注释库的正确方法